本章主要对circos环图中最基本的元素进行设置,包括karyotype、ideogram、ticks等模块。
Karyotype
Karyotype的信息就如同坐标轴一般,其大小、顺序、位置等直接决定了后续数据的展示。这里我们新建一个karyotype.conf文件用来设置karyotype的相关信息,主要设置的参数包括数据文件来源、是否使用特定染色体、染色体显示的单位大小(unit)、颜色、图中半径等等:
# 指定数据文件的位置,这里共使用了人类、小鼠、大鼠三组数据
karyotype = data/karyotype.human.txt,data/karyotype.mouse.txt,data/karyotype.rat.txt
# 使用哪些染色体,这里可以匹配正则表达式
chromosomes = -/[XY]/;-/rn/;-/mm/;rn1;mm1
# 染色体排序方式
chromosomes_order_by_karyotype = yes
# 染色体单位大小,在后续进行ticks等的设置时都会参考
chromosomes_units = 1000000
# 设置染色体颜色,不指定则用默认设置
chromosomes_color = /mm/:blues-5-seq-4;/rn/:reds-5-seq-4
# 将染色体反向排列
chromosomes_reverse = mm1
# 改变染色体在circos环图内所占比例
chromosomes_scale = rn1:0.25r;mm1:0.25r
# 改变染色体在circos环图内半径大小,如图中右上角
chromosomes_radius = hs2:1.05r;hs3:1.20r;hs4:1.35r;hs5:1.15r;hs6:1.05r
这时,我们的circos环图变成下图:
Ideogram
Ideogram主要是karyotype相关的其它一些基本显示信息的设置,包括线条颜色、粗细、间隔,标签位置、字体等,这里我们新建一个ideogram.conf文件用来管理ideogram的相关信息(我们把染色体条带的信息也一并在这里设置了):
<ideogram>
show = yes
# 线条粗细
thickness = 25p
# 颜色填充
fill = yes
fill_color = black
# 在circos环图中位置
radius = 0.80r
# 标签
show_label = yes
label_font = default
# label位置在圈外侧250像素,当前版本貌似只能让label位于统一位置
label_radius = dims(ideogram,radius_outer) + 250p
label_size = 24p
# label与karyotype平行
label_parallel = yes
# 显示染色体条带,条带信息已在karyotype文件中
show_bands = yes
fill_bands = yes
band_stroke_thickness = 0
band_stroke_color = black
band_transparency = 4
#两条染色体之间的间隔
<spacing>
default = 30u
# 改变特定染色体之间的间隔
<pairwise "/hs/ /hs/">
spacing = 0.5r
</pairwise>
</spacing>
</ideogram>
我们再来看一下新的circos环图:
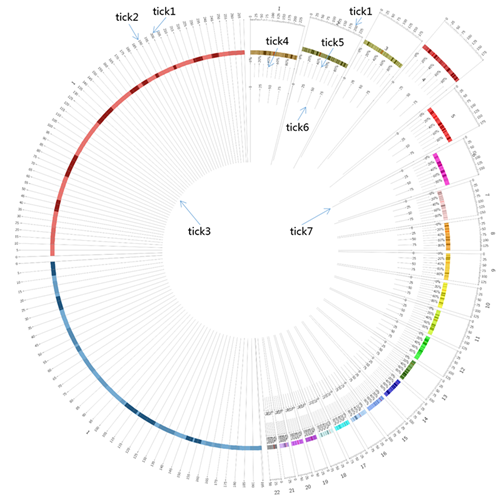
Ticks
Ticks主要是指坐标刻度和区块分隔线等,可以针对不同尺度设置不同的刻度显示方式。需要注意的是,Circos是由大至小进行刻度标识的,因此大尺度的刻度显示方式会替换小尺度。同样地,这里我们新建ticks.conf文件来设置相关信息:
show_ticks = yes
show_tick_labels = yes
show_grid = yes
# 全局设置
<ticks>
tick_label_font = light
# tick位置在外侧180像素,不同于label,tick能够跟随染色体位置而移动
radius = dims(ideogram,radius_outer) + 180p
label_offset = 5p
label_size = 16p
# 比例尺,这里指1代表1Mb
multiplier = 1e-6
color = black
thickness = 1p
# tick1
# u即karyotype中设置的chromosomes_units = 1000000,所以这里表示每25Mb处的刻度显示方式
<tick>
spacing = 25u
size = 12p
show_label = yes
format = %d
</tick>
# tick2
# 这里表示每5Mb处的刻度显示方式
<tick>
# 与其他tick标签至少保持1像素距离
# 由于Circos是由大至小进行刻度标识,所以该参数的设置使得在图中人类基因组的部分,这些刻度的标签不显示
label_separation = 1p
spacing = 5u
size = 7p
show_label = yes
format = %d
</tick>
# tick3
# 给小鼠和大鼠1号染色体设置grid
<tick>
# 说明该设置只限于特定染色体
chromosomes_display_default = no
chromosomes = rn1;mm1
spacing = 5u
# 这里由于spacing设置的是5u,所以此时size会被tick1和tick2替换,故无需设置
size = 0p
force_display = yes
# grid起始位置
grid_start = 0.45r
# grid终止位置,设置与外圈tick相同,使整体美观
grid_end = dims(ideogram,radius_outer) + 180p
grid_color = grey
grid_thickness = 1p
grid = yes
</tick>
# tick4
# 人类基因组每20%的相对比例刻度及grid
<tick>
# 不给小鼠和大鼠1号染色体设置
chromosomes = -rn1;-mm1
radius = 0.95r
# 说明该tick为相对比例刻度
spacing_type = relative
# 比例刻度为每20%
rspacing = 0.20
size = 6p
show_label = yes
# label为比例值(大小不超过1)
label_relative = yes
# 比例为百分比
rmultiplier = 100
format = %d
# label显示%作为后缀
suffix = %
# 为美观,不显示最后一个label
skip_last_label = yes
# 设置grid
grid_start = 0.885r
grid_end = 0.95r
grid_color = grey
grid_thickness = 1p
grid = yes
</tick>
# tick5
# 人类基因组每10%的相对比例刻度及grid,作为tick4的补充
<tick>
chromosomes = -rn1;-mm1
radius = 0.95r
spacing_type = relative
rspacing = 0.10
size = 3p
show_label = no
grid_start = 0.885r
grid_end = 0.95r
grid_color = lgrey
grid_thickness = 1p
grid = yes
</tick>
# tick6
# 人类基因组每25%的相对比例刻度及grid
<tick>
chromosomes = -rn1;-mm1
radius = 0.82r
spacing_type = relative
rspacing = 0.25
size = 6p
show_label = yes
label_relative = yes
rmultiplier = 100
format = %d
skip_last_label = yes
grid_start = 0.755r
grid_end = 0.82r
grid_color = grey
grid_thickness = 1p
grid = yes
</tick>
# tick7
# 利用相对比例刻度,为所有染色体首尾添加grid
<tick>
spacing_type = relative
rspacing = 1
# 为统一,显示一个刻度,同时为区别,大小和别的不一样
size = 6p
grid_start = 0.45r
grid_end = dims(ideogram,radius_outer) + 180p
grid_color = grey
grid_thickness = 1p
grid = yes
</tick>
</ticks>
现在,我们的circos环图显示了更多的信息: