由Martin Krzywinski等人开发的Circos,一出现便引起轰动,它打破了常规基因组学数据可视化的思路,通过对差异化交互数据的多维度展示,可以从不同层次全方位描述组学信息,让组学数据展示变成了艺术品。由于其实用性、美观性,Circos如今也广泛应用于社交网络、交通运输等领域。
 下面,我们就一步一步做出这张图(Circos包自带的demo)。
下面,我们就一步一步做出这张图(Circos包自带的demo)。
配置安装
最详尽的介绍和资料都在官网,可以在这里下载到最新的Circos软件包。我使用的版本号为0.69,操作系统为Ubuntu16.04(xenial,64bit),Windows下配置略有不同,可参考官方说明。
Circos本身是基于Perl的,所以不需要编译安装。解压下载的软件包到你想要存放的位置,并将circos/bin加入环境变量即可。通过circos -modules命令可以查看相关Perl模块的安装情况。Linux下CPAN可以通过$ sudo perl -MCPAN -e shell进入,然后执行install命令安装相应模块。容易出问题的主要是GD,在CPAN中直接安装GD可能出现错误:No success on command[/usr/bin/perl Build.PL –installdirs site],可以手动下载编译GD包,不过个人建议直接安装Ubuntu的libgd-perl包。
运行demo
我们需要认识到,Circos只是一个数据可视化工具,并不对数据本身进行分析处理。从其运行命令circos -conf circos.conf就可看出,所有的图形配置信息、数据文件信息等都可以保存在*.conf文件中。
如果直接运行circos命令,程序会在默认的路径下自动搜寻名为circos.conf的配置文件,可通过命令circos -debug_group io来查看程序默认的搜索路径,为避免错误,还是推荐主动指定配置文件。Circos包里有自己的demo,可通过circos -conf yourpath/circos/example/etc/circos.conf来运行,会在命令行当前目录下生成png和svg两个图形文件。
最简配置
配置文件中最少需要以下五个部分:
# Karyotypes
karyotype = data/karyotype.human.txt,data/karyotype.mouse.txt,data/karyotype.rat.txt
# Ideograms
<ideogram>
<spacing>
default = 0.002r
</spacing>
thickness = 25p
fill = yes
radius = 0.80r
</ideogram>
# Image
<image>
<<include etc/image.conf>>
</image>
# RGB/HSV color definitions, color lists, location of fonts, fill patterns.
<<include etc/colors_fonts_patterns.conf>>
# Debugging, I/O and other system parameters
<<include etc/housekeeping.conf>>
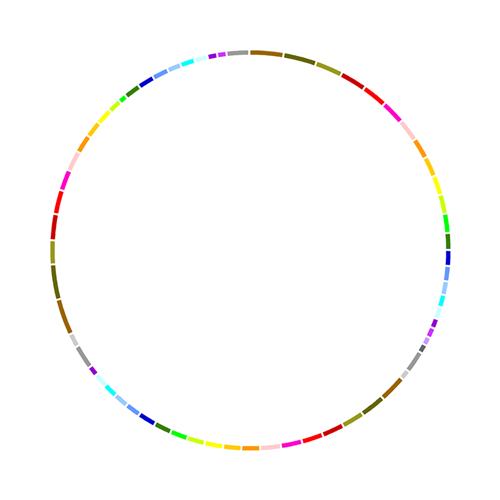
其中karyotype文件描述了染色体的名字、大小等基本信息,内容可以自主更改,不过这里我们先使用Circos默认提供的数据。后续我们还可以把karyotype、ideogram等分别放入各自的.conf文件中,通过<<include karyotype.conf>>等来引用,便于分类管理。现在,我们得到了下图,作为circos环图的最基本状态:
 除了上述,Circos中进行图层设置的还包含
除了上述,Circos中进行图层设置的还包含<ticks>模块来描述染色体上的刻度及区块,<zooms>模块来描述扩大或压缩的区域等;进行数据设置的还包含<highlights>标识高亮显示、<plots>描述数据图、<links>添加网络连线等;另外还有子模块<rules>描述注释规则、<backgrounds>描述背景图层、<axes>描述坐标轴等。
数据文件格式
Circos主要有四个模块需要输入数据文件,包括karyotype以及<highlight><plot><link>,格式要求都很简单,通过文本进行编辑即可。Circos默认使用空格作为分隔符,也可以在配置文件中通过命令file_delim = \t进行设置。
Karyotype可被视为circos环图的数据基础,描述了染色体的相关信息,其基本数据格式如下:
chr - hs1 1 0 249250621 chr1
chr - hs2 2 0 243199373 chr2
chr - hs3 3 0 198022430 chr3
...
需要注意的是,Circos使用前缀hs代表人类,而这里的chr1实际代表颜色(使用chr表示非物种特异)。同时还可以添加别的信息,如染色体条带等:
band hs1 p36.33 p36.33 0 2300000 gneg
band hs1 p36.32 p36.32 2300000 5400000 gpos25
band hs1 p36.31 p36.31 5400000 7200000 gneg
...
其它数据的格式主要有:
#chr start end [options]
hs5 50 75
#chr start end value [options]
hs5 50 75 0.75
hs5 50 75 ABC
[options]里的信息一般是<highlight> <plot> <link>里用到的变量或是颜色之类,如:
chr start end var1=value1,var2=value2,...
chr start end color=(R,G,B)
# 由于circos环图中元素众多,可能出现层次叠加问题,故可使用z值来界定,z值大的会覆盖在小的上面显示
chr start end z=1
对于链接数据links,格式有两种,一种是两个端点在同一行,即:
# chr1 start1 end1 chr2 start2 end2 [options]
hs1 200 300 hs10 1100 1300
hs7 50 150 hs 5000 6000 color=blue
另一种是通过唯一的id来标识两个端点:
# id chr start end [options]
link01 hs1 200 300
link01 hs2 1200 1300
link02 hs11 2200 2300
link02 hs21 4200 4300

