漫天繁星,你却只寻找那偏爱的motif…
一致性序列
如果基因组DNA是生命的设计图,那转录因子(再加上核酸酶等)就像小黄人一样,将各个重要部件解构出来。然而转录因子数量众多、功能不同,他们如何知道自己应该结合于基因组的位置,这就得依靠DNA序列motif来标识。下图即是CTCF的结合位点motif序列:
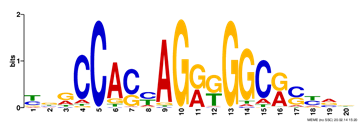 其中各个碱基高高低低的不同,反映了其出现的频率、保守性、对蛋白的亲和力等,并通过信息量(熵)的角度予以呈现(\(f_{b,i}\)表示碱基b在位置i出现的频率,\(p_b\)表示碱基b在基因组内的背景概率,通常要考虑保守性、GC含量等):
其中各个碱基高高低低的不同,反映了其出现的频率、保守性、对蛋白的亲和力等,并通过信息量(熵)的角度予以呈现(\(f_{b,i}\)表示碱基b在位置i出现的频率,\(p_b\)表示碱基b在基因组内的背景概率,通常要考虑保守性、GC含量等):
找到它们
通过motif序列,我们可以研究相应转录因子的功能,预测潜在的结合位点等等,因而搜索、计算出motif,成为转录因子研究的重要一步。
最直观的算法就是枚举,即把所有序列中的所有k-mer找出来,看看谁出现的次数最多。当然这样做对于数学家来说是不可接受的,于是有了诸如确定性最优化、概率最优化等方法,相关的软件也出了很多(上面的motif序列就是用MEME计算得到的),但谁才是最好的一直没有定论。也许是因为现在的数学模型对生物学问题的模拟还不够,不同的软件、算法总是对不同的数据集有效。对生物学家来说,目前的解决办法只能是:“多试试…”
反复出现必有因
Motif的概念并不局限于基因组DNA,RNA和蛋白质序列也可以抽提出相应的motif,重要的是我们如何理解其生物学意义。
解析序列信息,就像是解析一份密码、翻译一部外文书,大量重复出现的词语很多,身份意义却不同,有的可能起到结构性作用如“的地得”,有的指代明确如“中国美国俄罗斯”,有的则多词同义如“妈妈母亲额吉额娘”。从生物学的角度看待序列motif,就需要注意其生化特性、进化特点。比如转录因子的结合位点,其motif往往意味着某蛋白结构域与DNA碱基序列的相互作用,故有时需要特定的碱基组合,有时仅需要具相似生化特性的碱基组合。而同时,序列的进化保守性在其中的意义也需要进一步的思考,比如基因组内大量重复元件(repeat elements)存在的目的是什么,荒漠?残留?随机?结构组件?太多未知的因素需要探索,但存在即是合理,我们需要的是用更多角度来看待问题。

